AlphaGenome es un sofisticado modelo multimodal de aprendizaje profundo diseñado para decodificar el código regulador genético humano y murino mediante la predicción de datos genómicos funcionales a partir de secuencias de ADN. Procesa segmentos masivos de ADN de 1 Mb para generar predicciones de alta resolución en diversas categorías biológicas, como la expresión génica, los patrones de empalme y la accesibilidad de la cromatina. Al unificar el contexto de secuencias de largo alcance con la resolución de pares de bases, el modelo supera las desventajas tradicionales que anteriormente limitaban la genómica computacional. Una exhaustiva evaluación comparativa demuestra que AlphaGenome iguala o supera el rendimiento de las herramientas especializadas existentes en 25 de las 26 evaluaciones de predicción del efecto de las variantes. Este marco permite a los investigadores interpretar eficazmente cómo las mutaciones no codificantes influyen en los mecanismos moleculares y contribuyen a enfermedades complejas. En definitiva, el modelo proporciona una base de acceso abierto para la investigación genómica, agilizando la identificación de variaciones genéticas clínicamente relevantes.
Enlace al articulo cientifico, para aquellos interesados en profundizar en el tema: "Advancing regulatory variant effect prediction with AlphaGenome", por Žiga Avsec, y colegas. Publicado en Nature el 28 de Enero del 2026.
El resumen, la transcripción, y la traducción fueron hechas usando herramientas de software de Inteligencia Artificial.
El resumen se presenta en la forma de un diálogo entre dos personajes sintéticos que llamaremos Alicia y Beto.
Resumen
Beto
Sabes, estaba viendo algunos números antes de que empezáramos. Y hay una estadística en biología que siempre me impacta. Pasamos tanto tiempo hablando de los genes.
Alicia
Hmm, los planos.
Beto
Exacto. Los planos de todo: músculos, enzimas, color de ojos. Nos obsesionamos con ellos. Pero si miras realmente el genoma humano, todo eso solo constituye, ¿qué?, el 2% de nuestro ADN.
Alicia
Es un número realmente humillante, ¿no? Deja el 98% del genoma sin explicación. Al menos en la imaginación popular.
Beto
Claro. Y durante tanto tiempo los libros de texto simplemente se encogían de hombros y lo llamaban "ADN basura".
Alicia
Lo cual se siente un poco despectivo para algo que ocupa casi todo el espacio en la célula. “Basura” fue definitivamente la palabra equivocada, y costó caro.
Beto
Entonces, ¿qué es entonces?
Alicia
Ahora sabemos que es básicamente el sistema operativo. Es la materia oscura del genoma. Contiene todo el código regulador, los interruptores, los diales, los atenuadores, que le dicen a ese 2% cuándo activarse, dónde activarse y cuánto producir.
Beto
Vale, así que si los genes son las bombillas, esta materia oscura es todo el cableado y el tablero de interruptores oculto en la pared.
Alicia
Es una analogía perfecta.
Y eso nos lleva a la inmersión de hoy, porque nos hemos vuelto realmente buenos leyendo las bombillas. Sabemos cuándo un gen está roto, pero leer ese tablero de interruptores ha sido el desafío central de la biología moderna.
Beto
Porque hay millones de interruptores.
Alicia
Millones, y pequeños errores tipográficos en esa región no codificante, quiero decir, un solo cambio de letra puede causar enfermedades devastadoras: leucemia, autismo.
Beto
Pero, ¿cómo encuentras el único error tipográfico culpable entre millones de otros que son totalmente inofensivos?
Alicia
Es como tratar de encontrar un cable roto en todo el edificio Empire State con una linterna.
Beto
Bueno, parece que alguien acaba de inventar un reflector. Estamos viendo un artículo que acaba de salir en Nature hace un par de días, el 28 de enero de 2026. El título es “Advancing regulatory variant effect prediction with AlphaGenome”. Y la afirmación aquí es bastante audaz. Dice que este nuevo modelo de IA básicamente resuelve un compromiso fundamental que había frenado la genómica durante años.
Alicia
Es un salto enorme. Todo esto trata sobre AlphaGenome, este nuevo modelo unificado que han desarrollado. Y para entender por qué es algo tan grande, tenemos que hablar de ese compromiso que mencionaste. Porque hasta ahora los investigadores estaban atrapados entre dos opciones bastante imperfectas.
Beto
Bueno, desempaquémoslo. ¿Cuál era el estado del arte antes de AlphaGenome?
Alicia
Bueno, era el problema clásico de resolución versus contexto. Por un lado, tenías modelos especializados. Piénsalos como un microscopio súper potente. Podían hacer zoom sobre un tramo muy pequeño de ADN, quizá 10,000 letras, y decirte con precisión de una sola base si algo estaba mal justo ahí.
Beto
Así que podían detectar un error tipográfico, pero no podían ver la frase en la que estaba.
Alicia
Exacto. Visión de túnel total. Eran ciegos al paisaje más amplio. No podían decirte si un interruptor regulador a 50,000 letras de distancia intentaba controlar ese gen.
Beto
Y la otra opción eran los modelos generalistas.
Alicia
Esos eran como cámaras satelitales. Podían ver tramos enormes de ADN, cientos de miles de letras. Podían mapear todas esas conexiones a distancia, pero tenían visión borrosa.
Beto
¿Borrosa, cómo? Como baja resolución.
Alicia
Exactamente, baja resolución. Procesaban datos en “papeleras” (o "tarros"), agrupando, digamos, 128 letras de ADN juntas en un solo píxel. Así que podían decirte: “oye, hay algo mal en este vecindario”, pero no podían señalar la casa exacta.
Beto
Así que quedabas atrapado: un microscopio que no veía el panorama completo, o un mapa satelital demasiado pixelado para ser útil en un diagnóstico.
Alicia
Ese era el dilema. Y aquí está el momento “ajá” con AlphaGenome. Simplemente se niega a comprometerse. Procesa una cantidad masiva de contexto: una megabase. Eso es un millón de letras de secuencia de ADN a la vez.
Beto
Un millón de letras. Eso es cien veces más que esos modelos especializados.
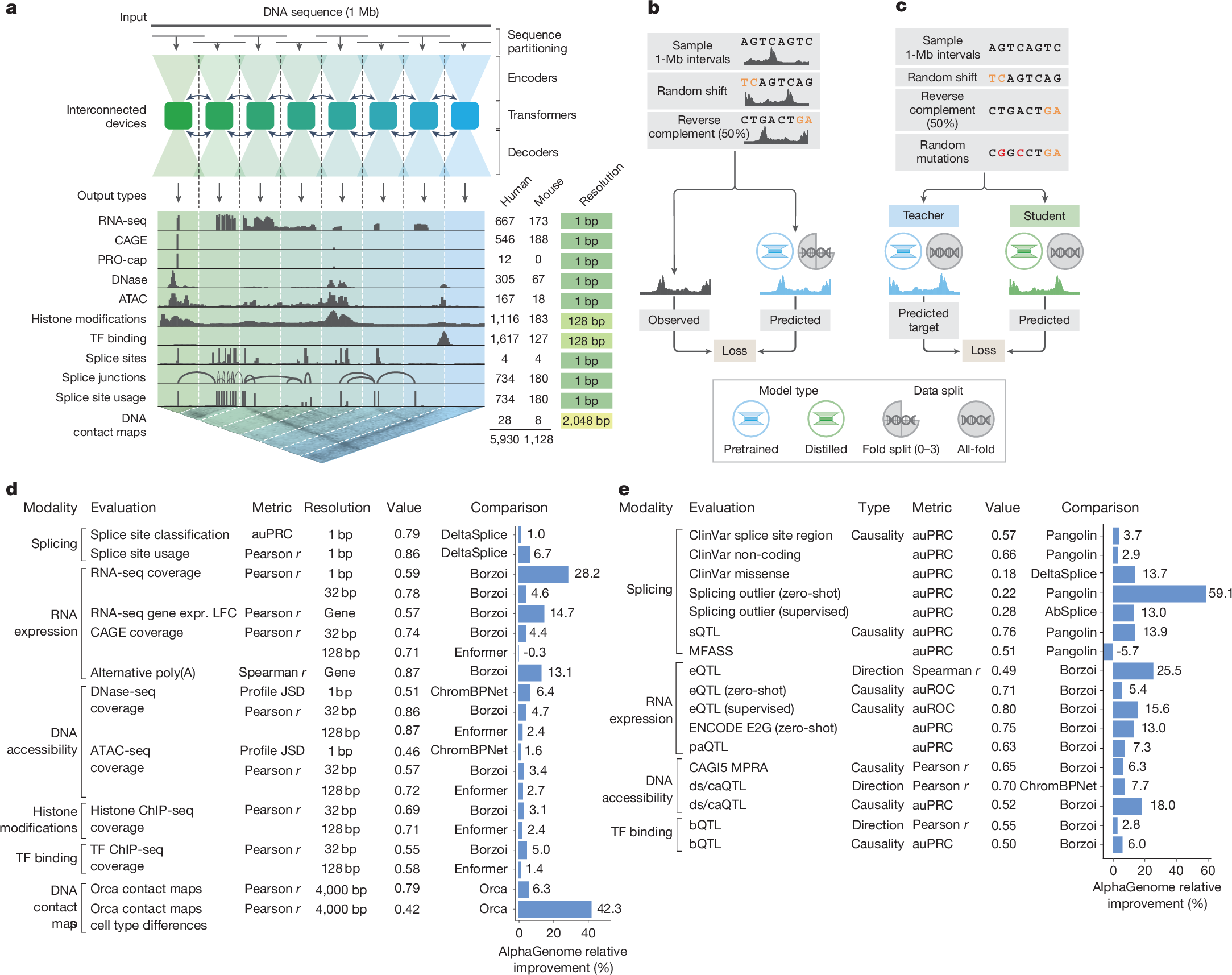
AlphaGenome. (a) Descripción general del modelo; (b) pre-entrenamiento; (c) destilación; (d, e) predicciones
Alicia
Es enorme. Pero, y esto es crucial, mantiene la resolución de una sola base. Te da la vista satelital de todo el continente, pero aún puedes hacer zoom hasta leer el número de la casa en el buzón.
Beto
Suena computacionalmente insano. Ya llegaremos a eso. Pero la biología no es solo una cadena de letras, ¿verdad? Es algo físico. Se pliega.
Alicia
Correcto. Y aquí es donde se vuelve como una navaja suiza. Es un modelo multimodal. No solo predice una cosa. Predice casi 6,000 características genómicas distintas al mismo tiempo.
Beto
6,000. ¿Qué tipo de características?
Alicia
Todo lo que un biólogo necesita. Predice la expresión génica: cuánto ARN se produce. Predice el splicing (empalme). El estado de la cromatina.
Beto
Rápida definición de estado de la cromatina.
Alicia
Piénsalo como accesibilidad. El ADN está enrollado alrededor de estos carretes de proteína. Si está demasiado apretado, el libro está cerrado. La maquinaria no puede leerlo. Si está suelto, el libro está abierto. AlphaGenome predice eso. Pero, honestamente, lo más impresionante es que predice la estructura 3D.
Beto
El plegamiento real del ADN.
Alicia
Sí. Dentro del núcleo, el ADN es una bola enmarañada de hilo. Un interruptor podría estar súper lejos de un gen si estiras el ADN en línea recta. Pero en ese enredo 3D pueden estar tocándose.
Beto
Y porque AlphaGenome mira esa ventana de un millón de letras, puede ver esos ciclos.
Alicia
Precisamente. De hecho, fíjate: el 99% de todas las conexiones conocidas entre potenciadores (enhancers) y genes ocurren dentro de esa ventana de una megabase. Así que al capturar ese alcance, captura el ciclo físico que conecta el interruptor con el gen. Está vinculando la geografía con la actividad.
Beto
Bien, tenemos un modelo que dice ver la imagen completa y los detalles mínimos. ¿Pero funciona? ¿O es el clásico “aprendiz de todo, maestro de nada”?
Alicia
Los datos dicen que es maestro de todo. Lo compararon con esos modelos súper especializados, los diseñados solo para splicing o solo para expresión. AlphaGenome igualó o superó a esos modelos en 25 de 26 evaluaciones.
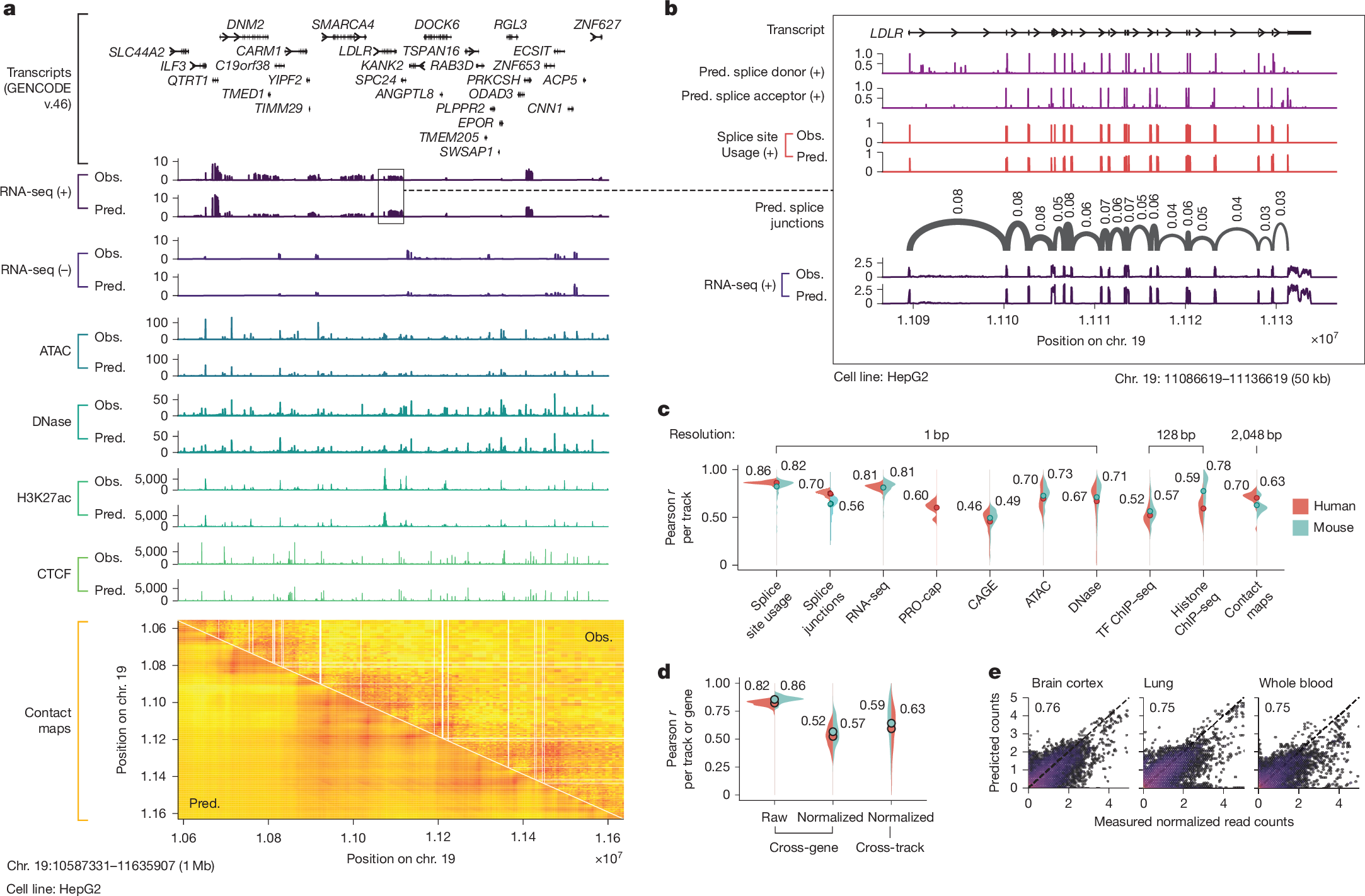
Ejemplo de predicción y desempeño de resultados
Beto
Vaya. Casi una barrida total.
Alicia
Y para entender por qué esto importa para nosotros deberíamos mirar una enfermedad específica. El artículo destaca el "splicing" como una gran victoria.
Beto
¿El splicing? Eso es el proceso de edición, ¿no? Como editar una película.
Alicia
Esa es la mejor analogía. Sí. Un gen se copia en ARN y es como una cinta cruda. Tiene escenas que quieres, los exones, y escenas que necesitas cortar, los intrones. El splicing es cortar los intrones y pegar los exones para hacer la película final.
Beto
Y si estropeas un corte, la proteína sale sin sentido.
Alicia
Completo sinsentido.
Y AlphaGenome es increíblemente bueno detectando malos cortes. Miraron un gen llamado DLG1 (sección b de la siguiente figura). Los investigadores habían encontrado una variante en el tejido de una arteria tibial de un paciente. Solo una pequeña deleción de cuatro letras.
Beto
Cuatro letras entre millones. Parece nada.
Alicia
Parece nada. Un modelo generalista con visión borrosa probablemente la pasaría por alto completamente. Pero AlphaGenome la vio y predijo el mecanismo biológico exacto.
Beto
¿Qué fue eso?
Alicia
Esas letras faltantes hicieron que la maquinaria celular omitiera un sitio de empalme. Predijo que esa escena se quedaría en la sala de montaje sin cortarse.
Beto
¿Y coincidió con lo que pasaba en el paciente?
Alicia
Perfectamente. Coincidió con la caída observada en la cobertura de ARN.
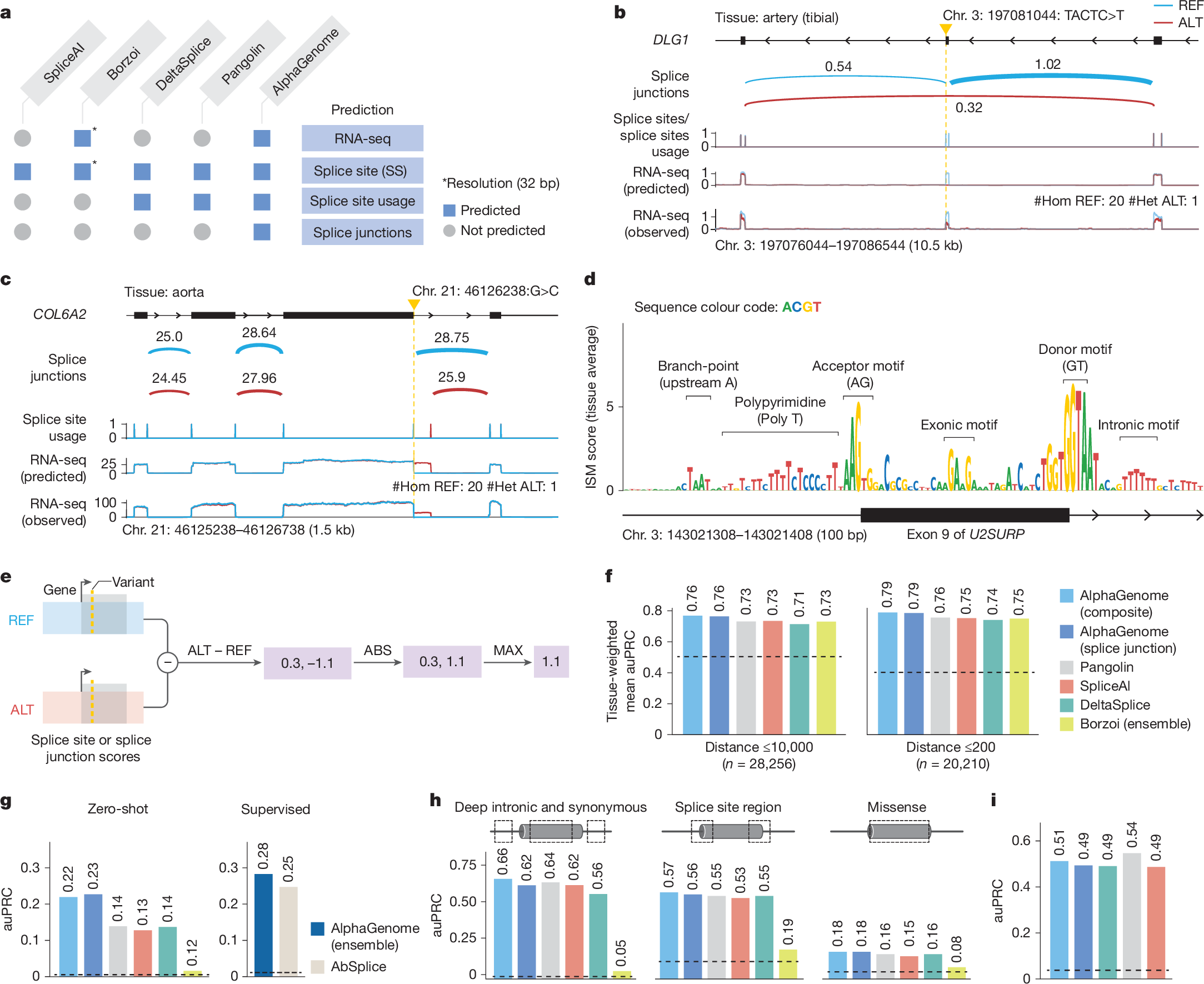
AlphaGenome es un modelo de predicción del efecto de la variante de empalme SOTA.
Pero también puede hacer lo contrario: predecir cuando una mutación añade un corte donde no debería haberlo.
Beto
Un neo-sitio de empalme.
Alicia
Exacto. Hubo un caso con el gen COL6A2 (sección c de la figura anterior). Una variante creó un nuevo sitio de empalme, profundo dentro de un intrón, allá en ese territorio que antes llamábamos basura. Las herramientas especializadas suelen perderlo porque solo lo ven como ruido. AlphaGenome lo marcó correctamente como un nuevo sitio donante que destrozaría la proteína.
Beto
Está capturando cosas que antes se escapaban por las grietas.
Pero, quiero llegar al ejemplo más potente del artículo. Hicieron un estudio de caso sobre leucemia que realmente parece conectar todos los puntos.
Alicia
Sí. El estudio de caso T-ALL, leucemia linfoblástica aguda T. Es un cáncer muy agresivo. A menudo está impulsado por un gen llamado TAL1.
Beto
TAL1 es un oncogén. ¿Qué significa?
Alicia
Es decir, promueve el crecimiento celular. En un adulto sano debería estar apagado. Pero si se enciende, si el acelerador se queda pillado, aparece el cáncer.
Beto
Entonces los investigadores encontraron una mutación cerca de este gen.
Alicia
Una pequeña inserción. Unas pocas letras extra en la región no codificante. Y aquí es donde AlphaGenome realmente brilla. No solo dijo “hmm, esto parece malo”. Predijo la formación de un neo-potenciador.
Beto
Un interruptor nuevo.
Alicia
Un interruptor nuevo que no existía antes. El modelo predijo un pico en marcas de histona, básicamente carteles de neón que se encienden en ese punto. Y, crucialmente, predijo que ese nuevo interruptor encendería el gen TAL1, que estaba a 7,500 bases de distancia.
Beto
Espera, 7.5 kilobases.
Alicia
Sí.

Interpretación de los efectos de las variantes en distintas modalidades con AlphaGenome. T-ALL y TAL1.
Beto
Si hubieras usado uno de esos modelos antiguos especializados con una ventana de, digamos, 10 kilobases, ...
Alicia
Podrías ver la mutación pero no ver el gen al que atacaba. Perderías la conexión completamente. Necesitas ese gran angular.
Beto
Y AlphaGenome vio la conexión.
Alicia
Lo vio. Pero luego los investigadores hicieron algo realmente interesante: le preguntaron a la IA por qué.
Beto
Esto fue la mutagénesis in silico.
Alicia
Sí. Básicamente interrogaron el modelo. Preguntaron: “vale, crees que esto enciende al gen, ¿pero qué letras específicas te hacen pensar eso?” Y el modelo reveló que la mutación había creado por accidente un motivo de unión perfecto, una plataforma de aterrizaje para una proteína llamada MYB.
Beto
¿MYB?
Alicia
Es un factor de transcripción, una proteína que flota por el núcleo buscando una secuencia de ADN específica para unirse. Es una llave buscando una cerradura.
Beto
Y la mutación construyó una cerradura.
Alicia
Construyó una cerradura perfecta para la llave MYB. Una vez que MYB se posó ahí, pum, encendió el gen de la leucemia.
Beto
Eso es aterradoramente mecánico.
Alicia
La biología es mecánica, y este modelo nos deja ver los planos. Transforma la IA de ser una caja negra que suelta una probabilidad a ser un verdadero generador de hipótesis. Le dice al médico: “este paciente tiene leucemia porque este cambio de letra específico reclutó la proteína MYB”. Eso es inteligencia accionable.
Beto
Eso nos lleva a otro gran problema en genética, ¿no? Una cosa es saber que una variante hace algo. Otra es saber si sube o baja un gen, la dirección del efecto.
Alicia
La direccionalidad es absolutamente crítica. Si estás diseñando un fármaco, necesitas saber si intentas bloquear una señal o potenciarla.
Beto
De nada sirve saber que la perilla de volumen está rota si no sabes si está atascada al máximo o en silencio.
Alicia
Exacto. Y los modelos anteriores eran francamente terribles en eso, básicamente una moneda al aire. Pero AlphaGenome ha mejorado significativamente esa predicción del signo. Lo probaron con eQTLs, que son ...
Beto
... los términos técnicos para variantes que cambian los niveles de expresión génica.
Alicia
Y esto tiene enormes implicaciones para los estudios de asociación del genoma (GWAS).
Beto
Siempre imagino un GWAS como una rueda de identificación policial. Ocurrió un crimen en un barrio. Tienes 10 sospechosos en fila, pero no sabes cuál lo hizo.
Alicia
Excelente analogía. GWAS te da ese conjunto creíble, esa nube de variantes que podrían ser la causa. Pero normalmente te quedas parado mirando la nube.
Beto
AlphaGenome despeja la niebla.
Alicia
Lo hace, de verdad. En el 49% de esos conjuntos, AlphaGenome pudo señalar con confianza al menos una variante y decir: “esta aumenta el gen” o “esta lo disminuye”.
Beto
49%. ¿Y antes?
Alicia
Con herramientas antiguas y puntuaciones conservadoras era alrededor del 11%.
Beto
Pasaste de resolver uno de cada diez casos a casi uno de cada dos. Eso es un salto enorme.
Alicia
Cambia la economía de la investigación. Pasas de investigar 10 callejones sin salida a enfocarte en el único culpable probable.
Beto
Quiero abrir el capó un segundo. ¿Cómo es esto siquiera posible? ¿Cómo ve un millón de letras a la vez con ese detalle? ¿Está corriendo en un superordenador del tamaño de un edificio?
Alicia
La arquitectura es en realidad muy ingeniosa. Combina dos tipos de redes neuronales que no suelen juntarse: una U-Net y un transformer.
Beto
Desglósalo para nosotros.
Alicia
Piensa en la U-Net como la parte que preserva el detalle. Esa es la componente microscopio, asegurándose de mantener la resolución de una sola base. El transformer es el mecanismo de atención. Es la parte que explora la secuencia de un millón de letras y dice: “oye, este fragmento de aquí es relevante para aquel fragmento allá”.
Beto
La U-Net maneja el detalle local y el transformer el contexto a largo alcance.
Alicia
Así es. Pero la verdadera salsa secreta estuvo en el entrenamiento. Usaron una técnica llamada destilación de conocimiento.
Beto
No tiene que ver con whiskey, al menos no aquí.
Alicia
En este caso, implica modelos profesor y alumno. Entrenan modelos profesor masivos, lentos y computacionalmente caros con enormes cantidades de datos. Luego entrenan un modelo alumno más pequeño para que imite a los profesores.
Beto
¿Y la ventaja del modelo alumno?
Alicia
Velocidad, pura velocidad. El modelo final puede analizar un millón de pares de bases y predecir las ~6,000 pistas en menos de un segundo en una sola GPU Nvidia H100.
Beto
Menos de un segundo.
Alicia
Sí. Lo que significa que realmente puedes ejecutarlo sobre millones de variantes en miles de personas. Hace posible el análisis a escala poblacional.
Beto
Increíble. Pero debo jugar al escéptico. Entonces, ¿la biología ya está resuelta? ¿Hemos terminado?
Alicia
Lejos de eso. Y los autores son muy honestos sobre las limitaciones. AlphaGenome es una herramienta asombrosa, pero no es una bola de cristal.
Beto
¿Cuáles son sus puntos ciegos?
Alicia
Bueno, una megabase es enorme, pero algunas interacciones ocurren a distancias aún mayores. Si un potenciador está a dos megabases de distancia, AlphaGenome todavía lo va a perder. Toca el borde del mapa.
Beto
Entonces la vista del satélite todavia tiene bordes.
Alicia
Sí. Y otra grande es el genoma personal. El modelo es genial mirando un genoma de referencia y evaluando una variante. Pero no ha sido probado rigurosamente sobre tu genoma específico con tu combinación de millones de variantes.
Beto
Porque mis variantes podrían interactuar entre sí de formas que el modelo no ha visto.
Alicia
Exacto. Es la diferencia entre detectar un error tipográfico en un diccionario y entender cómo ese error cambia la trama de una novela compleja. No hemos llegado a la etapa novela.
Beto
Y sigue estando mayormente enfocado en genes codificantes de proteínas y en humanos y ratones.
Alicia
Correcto. Si eres un veterinario, y trabajas con caballos o eres botánico, tendrás que esperar. Y aunque funciona bien con especificidad de tejido, aún lucha con algunas sutilezas que hacen diferente a una célula cerebral de una cardíaca.
Beto
Volviendo a la materia oscura, realmente se siente que AlphaGenome está encendiendo las luces en esa habitación oscura.
Alicia
Lo está. Piensa en ello como un laboratorio in silico. Antes, si encontrabas una variante en un paciente tenías que ir al laboratorio húmedo: pipetas, cultivar células, crear ratones transgénicos, tomar meses y costar una fortuna.
Beto
¿Y ahora?
Alicia
Ahora priorizas. Corres el genoma del paciente por el modelo. Te dice: "ignora estas 5,000 variantes, excepto esta. Esta crea una plataforma de unión para MYB y enciende un oncogén". Llevas solo esa al laboratorio.
Beto
Cambia la biología de ser puramente observacional a ser predictiva.
Alicia
Y eso lleva al pensamiento más emocionante para el futuro. Si podemos predecir el código, ...
Beto
... podemos escribirlo.
Alicia
Podemos escribirlo. Hablo de cribado virtual para biología sintética. Si realmente entendemos la gramática de esta materia oscura, si sabemos exactamente cómo construir un potenciador que solo active un gen en el hígado o solo en respuesta a cierta señal, podemos diseñar secuencias reguladoras personalizadas desde cero.
Beto
Así que en lugar de solo arreglar genes rotos, podemos diseñar sistemas de control enteramente nuevos.
Alicia
Exacto. Imagina una terapia génica que incluya un interruptor escrito a medida que el cuerpo del paciente reconoce perfectamente porque usamos AlphaGenome para verificar la gramática primero. Ya no solo leemos el sistema operativo.
Beto
Estamos obteniendo las herramientas para programarlo. Es un lugar salvaje en el que aterrizar. Hemos pasado de llamar “basura” al 98% de nuestro ADN a darnos cuenta de que es el software más sofisticado de la Tierra y ahora estamos aprendiendo a programarlo.
Alicia
Es un mundo nuevo y valiente, sin duda.
Beto
De verdad lo es. Gracias por guiarme a través de esto. Ha sido una inmersión profunda en AlphaGenome. Y honestamente, creo que no volveré a ver una hélice de ADN igual.
Alicia
Un placer.
Beto
Y a ti que nos escuchas, gracias por estar aquí. Si quieres profundizar en la arquitectura de la U-Net, y estudiar las gráficas sobre ese caso de leucemia, que recomiendo mucho, consulta la fuente. Nos vemos en el próximo análisis profundo.




